Le baromètre santé adulte est une enquête de grande échelle menée par l’Agence sanitaire et sociale de Nouvelle-Calédonie (ASS-NC). L’IPNC a participé à l’étude 2021-2022.
L’objectif initial de la participation de l’IPNC au baromètre santé adulte était de déterminer à quelles arboviroses ont été exposées les Calédoniens et en quelles proportions. Sur les 1800 logements visités dans le cadre de cette étude, 750 prises de sang ont été effectuées chez des Calédoniens de 18 à 64 ans sur l’ensemble du territoire, îles comprises. « Cela permet d’avoir une bonne estimation à l’échelle de la Nouvelle-Calédonie en incluant les trois Provinces », précise Myrielle Dupont-Rouzeyrol, responsable de l’unité de recherche et d’expertise dengue et arboviroses, en charge de cette enquête de séroprévalence.
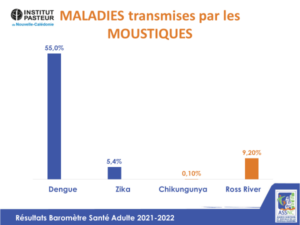
Arboviroses
Ainsi, 55% des adultes calédoniens testés ont déjà rencontré au moins une fois la dengue au cours de leur vie et présentent des anticorps résiduels. « Ce niveau est plutôt faible, en Polynésie française, par exemple, le pourcentage est plus proche de 80% », précise Myrielle Dupont-Rouzeyrol. Pour les autres arboviroses, l’immunité est nettement plus faible ; elle est inférieure à 10%. Il existe ainsi une certaine vulnérabilité à ces infections transmises par les moustiques. Ces résultats vont permettre d’ajuster les mesures de prévention de santé publique pour réduire le risque de transmission vectorielle. C’est la toute première fois que ces pourcentages sont calculés et présentés en Nouvelle-Calédonie.
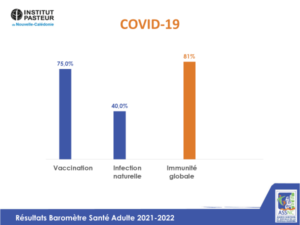
Covid-19
L’étude, débutée en août 2021, devait se concentrer sur les arbovirus. Or, l’arrivée du Covid-19 sur le territoire a modifié cet objectif initial. Un dépistage des anticorps de la Covid-19 a ainsi été ajouté afin d’obtenir une photographie à un instant t du pourcentage de Calédoniens exposés au virus. Au 31 juillet 2022, 81% des personnes testées présentaient des anticorps issus de la vaccination et/ou d’une infection naturelle. « On peut noter que 40% des individus avaient fait une infection récente ; ce qui permet d’estimer la circulation du virus à cette date, en pleine vague Omicron. » Les anticorps de la Covid-19 diminuant progressivement dans le temps en l’absence de stimulation, l’immunité devrait être plus faible aujourd’hui.

Projet dupliqué au Vanuatu
Cette même étude est également menée au Vanuatu depuis octobre 2022. Cette enquête de grande ampleur compte plus de 1000 participants de 5 à 85 ans et évalue l’immunité contre diverses maladies infectieuses dans la population du Vanuatu. Les dernières inclusions ont été effectuées en mai ; Myrielle Dupont-Rouzeyrol espère des résultats d’ici la fin de l’année.







