SARMPac : Surveillance des souches de Staphylococcus aureus résistantes à la méticilline dans le Pacifique
Contexte :
Staphylococcus aureus communément appelé Staphylocoque doré, est la principale espèce bactérienne responsable d’infections pouvant aller du simple furoncle à des pathologies bien plus graves (ostéomyélites, endocardites) voire mortelles (pneumopathies nécrosantes). Ceci est particulièrement le cas, dans les pays tropicaux, où le staphylocoque doré trouve des conditions climatiques (chaleur et humidité) idéales à son développement. Longtemps considéré comme un germe responsable d’infections nosocomiales (contractées à l’hôpital), ce dernier s’est aujourd’hui propagé en communautaire (en ville) où il représente un réel problème de santé publique. A cela s’ajoute, son niveau de résistance aux antibiotiques qui est en pleine ascension en Nouvelle-Calédonie, suspectant l’émergence d’un nouveau clone avec une production très fréquente de toxines entraînant des répercussions cliniques gravissimes.
Au travers du projet, nous proposons d’étudier les souches de S. aureus résistantes à la méticilline (SARM) circulantes dans le Pacifique afin de mieux comprendre les raisons de leur dissémination en milieu communautaire ceci dans le but d’enrayer ce phénomène.
Staphylococcus aureus résistant à la méticilline. (C)
Objectifs :
Les activités prévues dans le cadre de ce projet de santé publique visent à :
- actualiser les connaissances sur les souches de Staphylococcus aureus résistances à la méticilline circulantes dans le Pacifique
- améliorer et uniformiser les moyens de diagnostic microbiologique au sein des différents laboratoires partenaires.
Ce projet permettra aussi de renforcer les liens entre les différents laboratoires participants, d’améliorer les moyens de diagnostic biologique afin de mettre en place un système de surveillance optimal face à ce grave problème de santé publique.
Financement :
Ce projet est financé par le Fond Pacifique, le Centre National de références des Staphylocoques, le CHT et l’IPNC.
Méthodes :
La première étape a consisté à faire un état des lieux des techniques d’identification et d’antibiogramme dans les 5 laboratoires partenaires. La deuxième étape a été la réalisation d’une mission de formation et de transfert de compétences réalisées par les équipes de l’IPNC et du CHT. Ensuite, les inclusions des souches de SARM ont eu lieu en août-septembre 2019 (saison fraîche) et en janvier-février 2020 (saison chaude). Durant ces 2 périodes, toutes les souches de SARM acquises dans la communauté ont été incluses dans l’étude. Les données cliniques des patients (type d’infection, antécédents médicaux, traitement…) et les données relatives aux souches (type de résistance) ont été recueillies. Les souches ont été envoyées au CNR Staphylocoques qui a effectué la caractérisation moléculaire à l’aide de puces à ADN (S. aureus genotyping kit 2.0 Alere) ciblant 185 gènes, de nombreux facteurs de virulence (adhésines, exoprotéines, toxines) et la plupart des gènes de résistance aux antibiotiques. Le profil génétique a été recherché en déterminant le « Sequence Type » par Multilocus Sequence Typing (MLST) afin de caractériser les clones circulant dans le Pacifique.
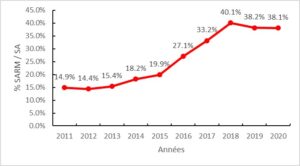
Evolution de la résistance à la méticilline au sein des S. aureus isolés en Nouvelle-Calédonie (Données CHT)
Résultats :
L’aperçu des techniques a été réalisé et a montré 4 méthodes d’identification et 3 méthodes d’antibiogramme différentes parmi les 5 laboratoires. Il a essentiellement mis en évidence les besoins de formation au Vanuatu. Ces formations et transferts de compétences ont eu lieu en juillet 2019 avant la collecte des souches. L’inclusion des souches a été réalisée : 300 SARM collectés en août/septembre 2019 : NC : 200, FP : 84, Fiji : 30, WF : 4, Vanuatu : 2. La caractérisation moléculaire a été réalisée sur plusieurs souches (NC : 49 ; FP : 84 et WF : 4). Des similitudes sont observées entre les trois territoires : une occurrence de SARM acquis dans la communauté PVL+. Cependant, il existe une dominance d’un clone particulier pour chaque territoire : en Nouvelle-Calédonie (CC6-MRSA-[IV+fus] (PVL+)), en Polynésie française (CC8-MRSA-IV(PVL+)) et à Wallis et Futuna (CC30-MRSA-IV (PVL+)). Compte tenu de la situation sanitaire, un travail spécifique sur les souches calédoniennes a été réalisé. L’étude a montré la dominance du clone résistant à l’acide fusidique (PVL+) associée à l’augmentation du Staphylococcus aureus résistant à la méticilline en Nouvelle-Calédonie. Sur la base de cette étude, le gouvernement de la Nouvelle-Calédonie a interdit la commercialisation de l’acide fusidique topique surutilisé sur le territoire ces dernières années. L’étude de la résistance à la mupirocine (pommade pour prévenir les infections par S. aureus) a montré que 4 souches résistantes / 200 souches NC.
Prespectives :
Les résultats générés par ce projet serviront de base aux études et aux actions de santé publique dans la région du Pacifique. En fonction des résultats, ce projet pourrait être étendu à d’autres pays du Pacifique.
SARMPac: Surveillance of methicillin-resistant Staphylococcus aureus (MRSA) strains in the Pacific
Context:
Staphylococcus aureus is the main bacterial specie responsible for infections ranging from simple boils to more serious diseases (osteomyelitis, endocarditis) and sometimes fatal (necrotizing pneumopathies). This is particularly the case in tropical countries, where this germ finds ideal climatic conditions for its development (heat and humidity). Long regarded as a germ responsible for nosocomial infections (contracted at the hospital), the latter has today spread in community (city) where it represents a real public health problem. In addition, its level of resistance to antibiotics is rising rapidly in New Caledonia, suggesting the emergence of a new clone with a very frequent production of toxins leading to serious clinical repercussions. We propose to study the methicillin-resistant S. aureus strains circulating in the Pacific to better understand the reasons for their dissemination in community and to rule on the hypothesis of the emergence of a new clone.
Objectives:
The activities planned in the framework of this public health project aim to:
– update knowledge on the strains of S. aureus resistant to methicillin circulating in the Pacific.
– improve and standardize the microbiological diagnosis within the various partner laboratories.
Fundings:
Pacific Funds, NRC of Staphylococcus, CHT, IPNC
Methods:
The first step was to make an overview of identification and antibiogram techniques in the 5 partner laboratories. The second step was training and skills transfer missions carried out by the IPNC and CHT teams. Then, the inclusions of the MRSA strains was carried out in August-September 2019 (cool season) and in January-February 2020 (hot season). During these 2 periods, all community-acquired MRSA strains were included in the study. Patient’s clinical data (infection type, medical history, treatment …) and strain data (resistance-type) were collected. The strains have sent to the NRC of Staphylococcus which carried out the molecular characterization using microarrays (S. aureus genotyping kit 2.0 Alere) targeting 185 genes, numerous virulence factors (adhesins, exoproteins, toxins …) and most of antibiotic resistance genes. The genetic profile has been done by determining the « Sequence Type » by Multilocus Sequence Typing (MLST) to characterize circulating clones in the Pacific.
Results:
The overview of the techniques was realized and has showed 4 different identification methods and 3 different antibiogram methods among the 5 laboratories. It highlighted essentially the training needs in Vanuatu. These training and transfer of skills have occurred in July in 2019 before the collection of the strains. Strains inclusion was realized: 300 MRSA collected during August/September 2019: NC: 200, FP: 84, Fiji: 30, WF: 4, Vanuatu: 2. Molecular characterization has been carried out on several strains (NC: 49; FP: 84 and WF: 4). Similitudes are observed between the three territories: Occurrence of PVL+ Community acquired MRSA. However, there is a dominance of a particular clone for each territory: in New Caledonia (CC6-MRSA-[IV+fus] (PVL+)), in French Polynesia (CC8-MRSA-IV(PVL+)) and in Wallis and Futuna (CC30-MRSA-IV (PVL+)). Given the sanitary situation, a specific work on the New Caledonian strains was carried out. Study has shown the dominance of fusidic acid-resistant (PVL+) clone associated with the increase in methicillin-resistant Staphylococcus aureus in New Caledonia. Based on this study, New Caledonia Government prohibited the marketing of topic fusidic acid overused in the territory in recent years. Study of the resistance to mupirocin (ointment to prevent infection by S. aureus) have shown that 4 resistant strains / 200 NC strains.
Perspectives:
The results generated by this project will serve as a basis for studies and public health actions in the Pacific region. Depending on the results, this project could be extended to other Pacific countries.

